
QSAR Aquatic Toxicity
순서
1. 데이터 셋 설명
2. 브루트 포스 접근
3. TPOT (Tree-based Pipeline Optimization Tool)
데이터 셋

데이터 셋 다운로드 링크 : https://archive.ics.uci.edu/ml/machine-learning-databases/00505/
2019년 09월 23일에 UCI Machine Learning Repository에 등록된 나름 최근에 등록된 데이터 셋입니다. 밀라노에 위치한 대학 QSAR 연구 그룹에서 제작된 데이터 셋입니다.
데이터 셋 정보를 보겠습니다.

데이터 셋 정보를 읽어보면 이 데이터 셋은 Daphnia Magna에 대한 acute toxicity (급성 수생 독성)을 예측하기 위해 908개 화학 물질 세트에 있는 물고기 Pimemphales promelas(잉어과의 물고기)에 대한 급성 수생 독성을 예측하기 위한 정량적 회귀 QSAR 모델을 개발하는 데 사용되었다.
데이터 셋의 열에 대한 정보가 있습니다.

8개의 분자 기술과 1개의 정량적인 어트리뷰트가 있습니다. 우리는 이번에 LC50의 값을 예측할 것입니다. 뿐만 아니라, 어떤 모델이 이 데이터 셋에 있어서 최적화된 모델인지까지 TPOT을 통해서 알아볼 것입니다. 사용할 모델 메소드들은 아래에 나열할 것이지만, 선형 회귀 (linear regression), 릿지 회귀 (ridge regression), 결정 나무 회귀 (decision tree regression), knn 회귀 (knn regression), 서포트 벡터 회귀 (svr regression), 랜덤 포레스트 회귀 (random forest regression), 몬드리안 포레스트 (mondrian forest regression), xgboost regression 모델을 적용해볼 것입니다.
실제 데이터가 어떻게 생겼는지 보겠습니다.

위에서 설명됐던 것과 같이 총 9개의 열이 있고, 마지막 열 (LC50)을 예측해야 합니다.
코드
코드는 Google Colab에서 진행했습니다. 가장 먼저 필요한 라이브러리들을 설치하고 import합니다.


그 다음, 데이터 셋에 없었던 칼럼 이름들을 설정해줍니다. 위에 Data set information를 보면 9개의 어트리뷰트에 대한 설명이 있습니다. 설명에

따라서 칼럼 이름을 설정해줍니다.
그리고 칼럼 이름 설정이 잘 되었는지 확인하는 출력 코드를 작성합니다.

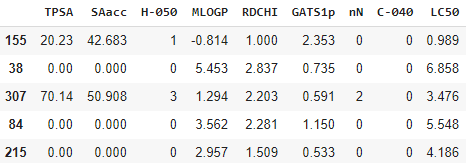
5개의 행을 랜덤하게 출력했지만, 알맞게 칼럼 이름들이 설정되었을 뿐만 아니라, 데이터 셋에도 blank나 NaN이 없는 것을 확인했습니다. 이제 데이터를 나누어 보겠습니다.
데이터를 아래와 같이 나누었습니다. y 변수는 데이터 셋의 LC50의 칼럼을 카피한 값만 가져갑니다. 왜냐하면 결국에 LC50의 값을 다양한 방법들을 통해서 예측할 것이기 때문입니다. 그리고 X는 데이터 셋에서 LC50 칼럼을 제외한 데이터를 저장하고 있습니다.

훈련 데이터, 테스트 데이터는 각 75%, 25%로 나눠주었습니다.
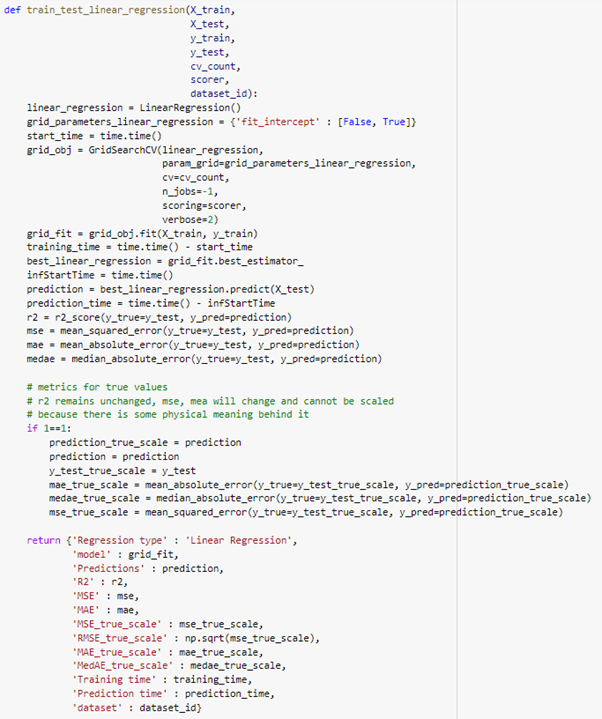
이제 아래부터는 예측을 위한 메소드들을 나열합니다. 물론 사이킷-런 (Sci-kit learn) 라이브러리만을 이용해서 할 수 있지만, 반환하는 값을 커스텀하게 해줄 것이기 때문에 직접 작성해야 했습니다. 한 번 모델을 이용하면 곧바로 반환하는 값으로 측정 메트릭과 예측값을 전달합니다.
- linear regression 선형 회귀 모델 메소드
- ridge regression 릿지 회귀 모델 메소드

- Decision tree 결정 나무 모델 메소드

- knn 회귀 모델 메소드

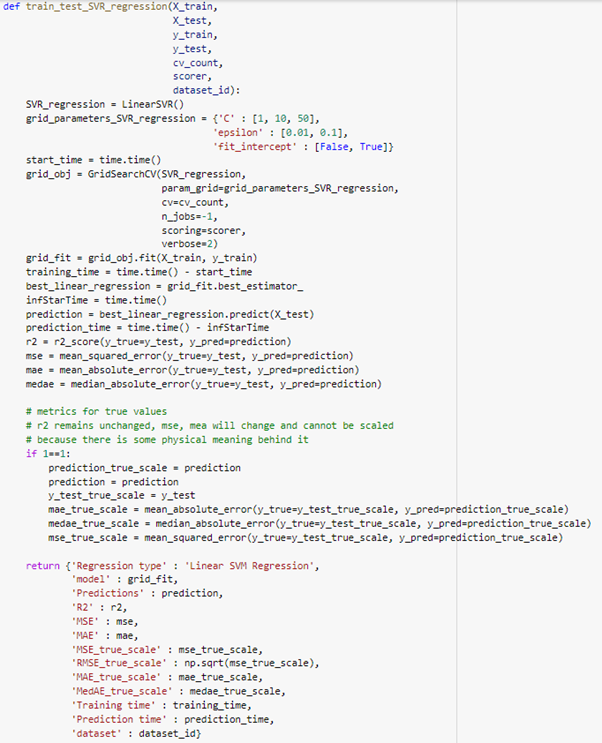
- SVR regression 서포트 벡터 회귀 모델 메소드
- 몬드리안 포레스트

- xgboost 회귀

모든 회귀 모델들을 메소드로 작성했습니다. 이제 메소드들에 패스될 변수들을 설정해줍니다. 그 이후에 실제로 메소드들에 데이터 셋을 넘겨서 LC50 예측값들을 확인해줍니다.
메소드들에 넘어갈 변수는 아래와 같이 설정해주었습니다. cv_count는 교차 검증을 얼만큼 할 것인가를 설정해줍니다. 그리고 scorer는 어떻게 메소드들을 평가할 것인지 메트릭의 한 종류를 설정해줍니다. 가장 무난한 mean_squared_error를 설정해줍니다. 그리고 results 딕셔너리는 (empty dictionary) 각 모델 메소드들의 결과값들을 딕셔너리에 모두 저장한 이후에 테이블로 출력하기 위함입니다.


데이터 셋을 모두 모델 메소드들에 패스시켜줍니다. 그리고 결과를 results 딕셔너리에 모두 저장함으로써 이후에 결과를 한 눈에 볼 수 있도록 합니다.
이제 메소드 별로 예측결과와 얼마나 효율이 좋은지 results를 테이블 형태로 출력함으로써 알아봅시다.

총 8개의 메소드들의 결과를 한 눈에 테이블로 확인할 수 있었습니다.
TPOT (Tree-based Pipeline Optimization Tool)
TPOT은 하이퍼 패러미터 튜닝과 feature selection을 자동으로 해주는 기술이라고 볼 수 있습니다. TPOT에 대한 자세한 설명은 코멘트에 달겠습니다.

이렇게 TPOT 라이브러리를 설치해줍니다.
이제 TPOTRegressor는 약 60분 동안 running하면서 어떤 모델이 적합한지, 어떤 하이퍼파라미터가 나은지, 모델의 메트릭 결과는 어떠한지 출력해줄 것입니다.
TPOTRegressor(
max_time_mins = 60 # 몇 분 동안 찾을 것인지 러닝타임을 분 단위로 설정해줄 수 있습니다.
verbosity = 2 # TPOT을 작동하고 얼마나 많은 정보를 출력할 것인지 정해줍니다. 0을 저장하면 아무것도 출력하지 않습니다. 1을 넣으면 최소한의 정보를 출력합니다. 2는 프로그레스바와 함께 1번보다는 많은 정보를 출력합니다. 3을 입력하면 모든 정보를 출력합니다.
n_jobs = -1 # TPOT 최적화 프로세스 동안 파이프라인을 평가하기 위해 병렬로 사용할 프로세스 수입니다. -1을 입력하면 동작하는 컴퓨터에서 가능한 모든 코어를 돌리겠다는 뜻입니다. )
이렇게 TPOT Regressor까지 동작해보았습니다. 모든 코드를 수행하는데 한 시간 넘게 걸렸습니다. 고생하셨습니다. 마지막으로 TPOT Regressor의 결과를 보겠습니다. 위 테이블에서도 모델 별 결과를 보면서 어떤 모델이 좋은지 확인할 수 있었습니다. 위 테이블을 다시 보면 Random Forest의 결과 메트릭 수치가 다른 모델들보다는 나은 것을 확인할 수 있었습니다.

약 60분 동안 검증을 43번 진행했습니다. 그리고 아래 보면 어떤 모델이 적합한지와 더불어서 괄호안에 하이퍼파라미터까지 결과로 알려줍니다. 정말 멋진 라이브러리가 아닐 수 없습니다.
이렇게 QSAR 수중 중독 LC50을 예측하고, TPOT (Tree-based Pipeline Optimization Tool)까지 알아보았습니다.
Whole Code
# QSAR Aquatic Toxicity
!pip install scikit-garden
import time
import pickle
import pandas as pd
import numpy as np
import seaborn as sns
import matplotlib.pyplot as plt
from sklearn.preprocessing import MaxAbsScaler
import sklearn.metrics
import xgboost
from sklearn.model_selection import train_test_split, GridSearchCV
from sklearn.metrics import r2_score, mean_squared_error, mean_absolute_error, mean_squared_log_error, median_absolute_error
from sklearn.ensemble import RandomForestRegressor
from sklearn.linear_model import LinearRegression, BayesianRidge
from sklearn.tree import DecisionTreeRegressor
from sklearn.neighbors import KNeighborsRegressor
from sklearn.svm import LinearSVR
from skgarden.mondrian import MondrianForestRegressor
columns = ["TPSA","SAacc", "H-050", "MLOGP", "RDCHI", "GATS1p", "nN", "C-040", "LC50"]
inputData = pd.read_csv("/content/qsar_aquatic_toxicity.csv",delimiter=";",names=columns)
display(inputData.sample(5))
display(inputData.describe())
y = inputData["LC50"].copy(deep=True)
X = inputData.copy(deep=True)
X.drop(["LC50"], inplace=True, axis=1)
scaler = MaxAbsScaler()
X_scaled = scaler.fit_transform(X)
X_train, X_test, y_train, y_test = train_test_split(X,y, test_size=0.25, random_state=42)
datasets = {}
datasets[0] = {'X_train': X_train,
'X_test' : X_test,
'y_train': y_train,
'y_test' : y_test}
def train_test_linear_regression(X_train,
X_test,
y_train,
y_test,
cv_count,
scorer,
dataset_id):
linear_regression = LinearRegression()
grid_parameters_linear_regression = {'fit_intercept' : [False, True]}
start_time = time.time()
grid_obj = GridSearchCV(linear_regression,
param_grid=grid_parameters_linear_regression,
cv=cv_count,
n_jobs=-1,
scoring=scorer,
verbose=2)
grid_fit = grid_obj.fit(X_train, y_train)
training_time = time.time() - start_time
best_linear_regression = grid_fit.best_estimator_
infStartTime = time.time()
prediction = best_linear_regression.predict(X_test)
prediction_time = time.time() - infStartTime
r2 = r2_score(y_true=y_test, y_pred=prediction)
mse = mean_squared_error(y_true=y_test, y_pred=prediction)
mae = mean_absolute_error(y_true=y_test, y_pred=prediction)
medae = median_absolute_error(y_true=y_test, y_pred=prediction)
# metrics for true values
# r2 remains unchanged, mse, mea will change and cannot be scaled
# because there is some physical meaning behind it
if 1==1:
prediction_true_scale = prediction
prediction = prediction
y_test_true_scale = y_test
mae_true_scale = mean_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
medae_true_scale = median_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
mse_true_scale = mean_squared_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
return {'Regression type' : 'Linear Regression',
'model' : grid_fit,
'Predictions' : prediction,
'R2' : r2,
'MSE' : mse,
'MAE' : mae,
'MSE_true_scale' : mse_true_scale,
'RMSE_true_scale' : np.sqrt(mse_true_scale),
'MAE_true_scale' : mae_true_scale,
'MedAE_true_scale' : medae_true_scale,
'Training time' : training_time,
'Prediction time' : prediction_time,
'dataset' : dataset_id}
def train_test_bRidge_regression(X_train,
X_test,
y_train,
y_test,
cv_count,
scorer,
dataset_id):
bRidge_regression = BayesianRidge()
grid_parameters_BayesianRidge_regression = {'fit_intercept' : [False, True],
'n_iter':[300,1000,5000]}
start_time = time.time()
grid_obj = GridSearchCV(bRidge_regression,
param_grid=grid_parameters_BayesianRidge_regression,
cv=cv_count,
n_jobs=-1,
scoring=scorer,
verbose=2)
grid_fit = grid_obj.fit(X_train, y_train)
training_time = time.time() - start_time
best_linear_regression = grid_fit.best_estimator_
infStartTime = time.time()
prediction = best_linear_regression.predict(X_test)
prediction_time = time.time() - infStartTime
r2 = r2_score(y_true=y_test, y_pred=prediction)
mse = mean_squared_error(y_true=y_test, y_pred=prediction)
mae = mean_absolute_error(y_true=y_test, y_pred=prediction)
medae = median_absolute_error(y_true=y_test, y_pred=prediction)
if 1==1:
prediction_true_scale = prediction
prediction = prediction
y_test_true_scale = y_test
mae_true_scale = mean_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
medae_true_scale = median_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
mse_true_scale = mean_squared_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
return {'Regression type' : 'Bayesian Ridge Regression',
'model' : grid_fit,
'Predictions' : prediction,
'R2' : r2,
'MSE' : mse,
'MAE' : mae,
'MSE_true_scale' : mse_true_scale,
'RMSE_true_scale' : np.sqrt(mse_true_scale),
'MAE_true_scale' : mae_true_scale,
'MedAE_true_scale' : medae_true_scale,
'Training time' : training_time,
'Prediction time' : prediction_time,
'dataset' : dataset_id}
def train_test_decision_tree_regression(X_train,
X_test,
y_train,
y_test,
cv_count,
scorer,
dataset_id):
decision_tree_regression = DecisionTreeRegressor(random_state=42)
grid_parameters_decision_tree_regression = {'max_depth' : [None, 3,5,7,9,10,11]}
start_time = time.time()
grid_obj = GridSearchCV(decision_tree_regression,
param_grid=grid_parameters_decision_tree_regression,
cv=cv_count,
n_jobs=-1,
scoring=scorer,
verbose=2)
grid_fit = grid_obj.fit(X_train, y_train)
training_time = time.time() - start_time
best_linear_regression = grid_fit.best_estimator_
infStartTime = time.time()
prediction = best_linear_regression.predict(X_test)
prediction_time = time.time() - infStartTime
r2 = r2_score(y_true=y_test, y_pred=prediction)
mse = mean_squared_error(y_true=y_test, y_pred=prediction)
mae = mean_absolute_error(y_true=y_test, y_pred=prediction)
medae = median_absolute_error(y_true=y_test, y_pred=prediction)
if 1==1:
prediction_true_scale = prediction
prediction = prediction
y_test_true_scale = y_test
mae_true_scale = mean_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
medae_true_scale = median_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
mse_true_scale = mean_squared_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
return {'Regression type' : 'Decision Tree Regression',
'model' : grid_fit,
'Predictions' : prediction,
'R2' : r2,
'MSE' : mse,
'MAE' : mae,
'MSE_true_scale' : mse_true_scale,
'RMSE_true_scale' : np.sqrt(mse_true_scale),
'MAE_true_scale' : mae_true_scale,
'MedAE_true_scale' : medae_true_scale,
'Training time' : training_time,
'Prediction time' : prediction_time,
'dataset' : dataset_id}
def train_test_knn_regression(X_train, X_test, y_train,
y_test,
cv_count,
scorer,
dataset_id):
knn_regression = KNeighborsRegressor()
grid_parameters_knn_regression = {'n_neighbors' : [1,2,3],
'weights': ['uniform', 'distance'],
'algorithm': ['ball_tree', 'kd_tree'],
'leaf_size': [30,90,100,110],
'p': [1,2]}
start_time = time.time()
grid_obj = GridSearchCV(knn_regression,
param_grid=grid_parameters_knn_regression,
cv=cv_count,
n_jobs=-1,
scoring=scorer,
verbose=2)
grid_fit = grid_obj.fit(X_train, y_train)
training_time = time.time() - start_time
best_linear_regression = grid_fit.best_estimator_
infStartTime = time.time()
prediction = best_linear_regression.predict(X_test)
prediction_time = time.time() - infStartTime
r2 = r2_score(y_true=y_test, y_pred=prediction)
mse = mean_squared_error(y_true=y_test, y_pred=prediction)
mae = mean_absolute_error(y_true=y_test, y_pred=prediction)
medae = median_absolute_error(y_true=y_test, y_pred=prediction)
# metrics for true values
# r2 remains unchanged, mse, mea will change and cannot be scaled
# because there is some physical meaning behind it
if 1==1:
prediction_true_scale = prediction
prediction = prediction
y_test_true_scale = y_test
mae_true_scale = mean_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
medae_true_scale = median_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
mse_true_scale = mean_squared_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
return {'Regression type' : 'KNN Regression',
'model' : grid_fit,
'Predictions' : prediction,
'R2' : r2,
'MSE' : mse,
'MAE' : mae,
'MSE_true_scale' : mse_true_scale,
'RMSE_true_scale' : np.sqrt(mse_true_scale),
'MAE_true_scale' : mae_true_scale,
'MedAE_true_scale' : medae_true_scale,
'Training time' : training_time,
'Prediction time' : prediction_time,
'dataset' : dataset_id}
def train_test_SVR_regression(X_train,
X_test,
y_train,
y_test,
cv_count,
scorer,
dataset_id):
SVR_regression = LinearSVR()
grid_parameters_SVR_regression = {'C' : [1, 10, 50],
'epsilon' : [0.01, 0.1],
'fit_intercept' : [False, True]}
start_time = time.time()
grid_obj = GridSearchCV(SVR_regression,
param_grid=grid_parameters_SVR_regression,
cv=cv_count,
n_jobs=-1,
scoring=scorer,
verbose=2)
grid_fit = grid_obj.fit(X_train, y_train)
training_time = time.time() - start_time
best_linear_regression = grid_fit.best_estimator_
infStarTime = time.time()
prediction = best_linear_regression.predict(X_test)
prediction_time = time.time() - infStarTime
r2 = r2_score(y_true=y_test, y_pred=prediction)
mse = mean_squared_error(y_true=y_test, y_pred=prediction)
mae = mean_absolute_error(y_true=y_test, y_pred=prediction)
medae = median_absolute_error(y_true=y_test, y_pred=prediction)
# metrics for true values
# r2 remains unchanged, mse, mea will change and cannot be scaled
# because there is some physical meaning behind it
if 1==1:
prediction_true_scale = prediction
prediction = prediction
y_test_true_scale = y_test
mae_true_scale = mean_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
medae_true_scale = median_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
mse_true_scale = mean_squared_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
return {'Regression type' : 'Linear SVM Regression',
'model' : grid_fit,
'Predictions' : prediction,
'R2' : r2,
'MSE' : mse,
'MAE' : mae,
'MSE_true_scale' : mse_true_scale,
'RMSE_true_scale' : np.sqrt(mse_true_scale),
'MAE_true_scale' : mae_true_scale,
'MedAE_true_scale' : medae_true_scale,
'Training time' : training_time,
'Prediction time' : prediction_time,
'dataset' : dataset_id}
def train_test_random_forest_regression(X_train,
X_test,
y_train,
y_test,
cv_count,
scorer,
dataset_id):
random_forest_regression = RandomForestRegressor(random_state=42)
grid_parameters_random_forest_regression = {'n_estimators' : [3,5,10,15,18],
'max_depth' : [None, 2,3,5,7,9]}
start_time = time.time()
grid_obj = GridSearchCV(random_forest_regression,
param_grid=grid_parameters_random_forest_regression,
cv=cv_count,
n_jobs=-1,
scoring=scorer,
verbose=2)
grid_fit = grid_obj.fit(X_train, y_train)
training_time = time.time() - start_time
best_linear_regression = grid_fit.best_estimator_
infStartTime = time.time()
prediction = best_linear_regression.predict(X_test)
prediction_time = time.time() - infStartTime
r2 = r2_score(y_true=y_test, y_pred=prediction)
mse = mean_squared_error(y_true=y_test, y_pred=prediction)
mae = mean_absolute_error(y_true=y_test, y_pred=prediction)
medae = median_absolute_error(y_true=y_test, y_pred=prediction)
# metrics for true values
# r2 remains unchanged, mse, mea will change and cannot be scaled
# because there is some physical meaning behind it
if 1==1:
prediction_true_scale = prediction
prediction = prediction
y_test_true_scale = y_test
mae_true_scale = mean_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
medae_true_scale = median_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
mse_true_scale = mean_squared_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
return {'Regression type' : 'Random Forest Regression',
'model' : grid_fit,
'Predictions' : prediction,
'R2' : r2,
'MSE' : mse,
'MAE' : mae,
'MSE_true_scale' : mse_true_scale,
'RMSE_true_scale' : np.sqrt(mse_true_scale),
'MAE_true_scale' : mae_true_scale,
'MedAE_true_scale' : medae_true_scale,
'Training time' : training_time,
'Prediction time' : prediction_time,
'dataset' : dataset_id}
def train_test_mondrian_forest_regression(X_train,
X_test,
y_train,
y_test,
cv_count,
scorer,
dataset_id):
mondrian_forest_regression = MondrianForestRegressor(random_state=42)
grid_parameters_mondrian_forest_regression = {'n_estimators' : [3,5,10,15,18,100],
'max_depth' : [None, 2,3,5,7,9,25,50]}
start_time = time.time()
grid_obj = GridSearchCV(mondrian_forest_regression,
param_grid=grid_parameters_mondrian_forest_regression,
cv=cv_count,
n_jobs=-1,
scoring=scorer,
verbose=2)
grid_fit = grid_obj.fit(X_train, y_train)
training_time = time.time() - start_time
best_linear_regression = grid_fit.best_estimator_
infStartTime = time.time()
prediction = best_linear_regression.predict(X_test)
prediction_time = time.time() - infStartTime
r2 = r2_score(y_true=y_test, y_pred=prediction)
mse = mean_squared_error(y_true=y_test, y_pred=prediction)
mae = mean_absolute_error(y_true=y_test, y_pred=prediction)
medae = median_absolute_error(y_true=y_test, y_pred=prediction)
# metrics for true values
# r2 remains unchanged, mse, mea will change and cannot be scaled
# because there is some physical meaning behind it
if 1==1:
prediction_true_scale = prediction
prediction = prediction
y_test_true_scale = y_test
mae_true_scale = mean_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
medae_true_scale = median_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
mse_true_scale = mean_squared_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
return {'Regression type' : 'Mondrian Forest Regression',
'model' : grid_fit,
'Predictions' : prediction,
'R2' : r2,
'MSE' : mse,
'MAE' : mae,
'MSE_true_scale' : mse_true_scale,
'RMSE_true_scale' : np.sqrt(mse_true_scale),
'MAE_true_scale' : mae_true_scale,
'MedAE_true_scale' : medae_true_scale,
'Training time' : training_time,
'Prediction time' : prediction_time,
'dataset' : dataset_id}
def xgboost_regression(X_train, X_test, y_train, y_test,
cv_count,
scorer,
dataset_id):
x_gradient_boosting_regression = xgboost.XGBRegressor(random_state=42)
grid_parameters_x_gradient_boosting_regression = {'n_estimators' : [3,5,10,15,50,60,80,100,200,300],
'max_depth' : [1,2, 3,5,7,9,10,11,15],
'learning_rate' :[ 0.0001, 0.001, 0.01, 0.1, 0.15, 0.2, 0.8, 1.0],
}
start_time = time.time()
grid_obj = GridSearchCV(x_gradient_boosting_regression,
param_grid=grid_parameters_x_gradient_boosting_regression,
cv=cv_count,
n_jobs=-1,
scoring=scorer,
verbose=2)
grid_fit = grid_obj.fit(X_train, y_train)
training_time = time.time() - start_time
best_linear_regression = grid_fit.best_estimator_
infStartTime = time.time()
prediction = best_linear_regression.predict(X_test)
prediction_time = time.time() - infStartTime
r2 = r2_score(y_true=y_test, y_pred=prediction)
mse = mean_squared_error(y_true=y_test, y_pred=prediction)
mae = mean_absolute_error(y_true=y_test, y_pred=prediction)
medae = median_absolute_error(y_true=y_test, y_pred=prediction)
# metrics for true values
# r2 remains unchanged, mse, mea will change and cannot be scaled
# because there is some physical meaning behind it
if 1==1:
prediction_true_scale = prediction
prediction = prediction
y_test_true_scale = y_test
mae_true_scale = mean_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
medae_true_scale = median_absolute_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
mse_true_scale = mean_squared_error(y_true=y_test_true_scale, y_pred=prediction_true_scale)
return {'Regression type' : 'XGBoost Regression',
'model' : grid_fit,
'Predictions' : prediction,
'R2' : r2,
'MSE' : mse,
'MAE' : mae,
'MSE_true_scale' : mse_true_scale,
'RMSE_true_scale' : np.sqrt(mse_true_scale),
'MAE_true_scale' : mae_true_scale,
'MedAE_true_scale' : medae_true_scale,
'Training time' : training_time,
'Prediction time' : prediction_time,
'dataset' : dataset_id}
# make scorer
scorer = 'neg_mean_squared_error'
results = {}
counter = 0
cv_count = 5
for dataset in [0]:
X_train, X_test, y_train, y_test = datasets[dataset]['X_train'], datasets[dataset]['X_test'], datasets[dataset]['y_train'], datasets[dataset]['y_test']
results[counter] = train_test_linear_regression(X_train, X_test, y_train, y_test, cv_count, scorer, dataset)
print("Linear Regression completed")
counter += 1
results[counter] = train_test_bRidge_regression(X_train, X_test, y_train, y_test, cv_count, scorer, dataset)
print("Bayesian Ridge Regression completed")
counter += 1
results[counter] = train_test_decision_tree_regression(X_train, X_test, y_train, y_test, cv_count, scorer, dataset)
print("Decision Trees completed")
counter += 1
results[counter] = train_test_knn_regression(X_train, X_test, y_train,y_test, cv_count, scorer, dataset)
print("KNN completed")
counter += 1
results[counter] = train_test_SVR_regression(X_train, X_test, y_train, y_test, cv_count, scorer, dataset)
print("SVR completed")
counter += 1
results[counter] = train_test_random_forest_regression(X_train, X_test, y_train, y_test, cv_count, scorer, dataset)
print("Random Forest completed")
counter += 1
results[counter] = train_test_mondrian_forest_regression(X_train, X_test, y_train, y_test, cv_count, scorer, dataset)
print("Mondrian Forest completed")
counter += 1
results[counter] = xgboost_regression(X_train, X_test, y_train, y_test, cv_count, scorer, dataset)
print("XGBoost completed")
counter += 1
results_df = pd.DataFrame.from_dict(results, orient='index')
display(results_df)
results_df.to_csv('results_df_manual.csv')
pickle.dump(results, open('results_manual.p', 'wb'))
!pip install tpot
from sklearn.model_selection import train_test_split
from sklearn.metrics import mean_squared_log_error, mean_squared_error, mean_absolute_error
columns = ["TPSA","SAacc", "H-050", "MLOGP", "RDCHI", "GATS1p", "nN", "C-040", "LC50"]
input_data = pd.read_csv("/content/qsar_aquatic_toxicity.csv",delimiter=";",names=columns)
# split data into X and y
from tpot import TPOTRegressor
y = input_data["LC50"].copy(deep=True)
X = input_data.copy(deep=True)
X.drop(["LC50"], inplace=True, axis=1)
X_train, X_test, y_train, y_test = train_test_split(X,
y,
test_size=0.25,
shuffle=True,
random_state=42)
tpot = TPOTRegressor(max_time_mins=60, verbosity=2, n_jobs=-1)
tpot.fit(X_train,y_train)
tpot.export('QSAR_aquatic_toxicity.py')
y_predictions = tpot.predict(X_test)
r2 = sklearn.metrics.r2_score(y_test, y_predictions)
mae = sklearn.metrics.mean_absolute_error(y_test, y_predictions)
mse = sklearn.metrics.mean_squared_error(y_test, y_predictions)
rmse = np.sqrt(mse)
print("R2 score:", r2)
print("MAE:", mae)
print("MSE:", mse)
print("RMSE:", rmse)
from sklearn.decomposition import FastICA
from sklearn.model_selection import train_test_split
from sklearn.pipeline import make_pipeline, make_union
from sklearn.preprocessing import Binarizer, MaxAbsScaler
from tpot.builtins import OneHotEncoder, StackingEstimator
from sklearn.preprocessing import FunctionTransformer
# NOTE: Make sure that the class is labeled 'target' in the data file
tpot_data = pd.read_csv('PATH/TO/DATA/FILE', sep='COLUMN_SEPARATOR', dtype=np.float64)
features = tpot_data.drop('target', axis=1).values
training_features, testing_features, training_target, testing_target = \
train_test_split(features, tpot_data['target'].values, random_state=None)
# Average CV score on the training set was:-0.9705966623633835
exported_pipeline = make_pipeline(
make_union(
make_pipeline(
make_union(
FunctionTransformer(copy),
FastICA(tol=0.35000000000000003)
),
MaxAbsScaler()
),
make_union(
FunctionTransformer(copy),
Binarizer(threshold=0.9500000000000001)
)
),
OneHotEncoder(minimum_fraction=0.05, sparse=False, threshold=10),
XGBRegressor(learning_rate=0.1, max_depth=9, min_child_weight=2, n_estimators=100, nthread=1, subsample=0.8500000000000001)
)
exported_pipeline.fit(training_features, training_target)
results = exported_pipeline.predict(testing_features)
'공부 > 인공지능' 카테고리의 다른 글
| 블로그 댓글 개수 예측하기(using Random Forest) (0) | 2021.01.13 |
|---|---|
| 커리어넷 대학정보 크롤링하기 (feat. Python Selenium) (0) | 2020.12.30 |
| 파킨스 텔레모니터링 데이터 셋 분석과 모델 훈련 (feat. 랜덤 포레스트) (1) | 2020.12.23 |
| BlogFeedback 피쳐셀렉션 (Feature Selection) (feat. Lasso, Ridge) (0) | 2020.12.21 |
| ML/DL for Everyone with PyTorch (Sung Kim) (0) | 2020.12.13 |



댓글